Transcriptomics (RNA-Seq)
Standard RNA sequencing
Standard RNA-seq(standard RNA sequencing)은 세포나 조직에서 추출한 mRNA를 Reverse transcription하여 cDNA로 변환한 뒤, Next-Generation Sequencing, NGS 기술로 분석해 유전자 발현을 정확화하는 방법입니다.
Service Features
| Platform (Library) | Illumina Novaseq (PE150) DNBSEQ-T7 |
|---|---|
| Sample Type | RNA |
| Sequencing Data | 1-2 Gb for Prokaryotes, 6-10 Gb for Eukaryotes |
| Turnaround Time | ~4 weeks after sample QC |
| Analyzable Species | Prokaryotes (Standard or rRNA depleted directional library) Eukaryotes (Poly A enriched, Tissues and Plants) |
*RNA prep. 서비스가 필요하신 고객은 상담을 통해 진행 가능합니다.
Service Progress
Data Analysis
RNA sequencing을 통해 얻은 수백만 개의 Short reads는 Reference genome에 정렬되어 각 유전자의 발현량을 계산하거나, 새로운 Transcriptome, Alternative splicing, Gene fusion 등을 탐지할 수 있습니다. Illumina 플랫폼을 사용하며, DEG 탐색, Pathway analysis 등에 폭넓게 활용됩니다.
Demo Results
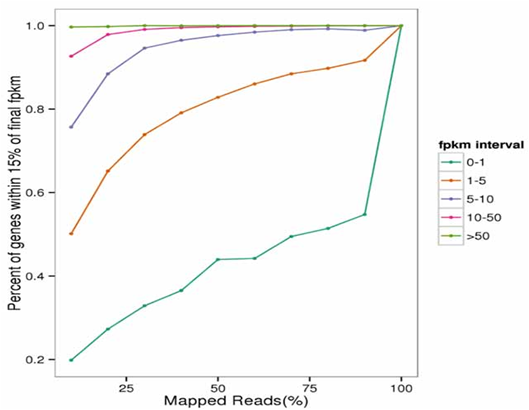
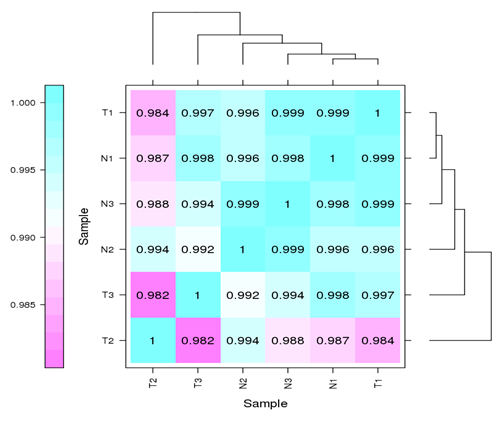
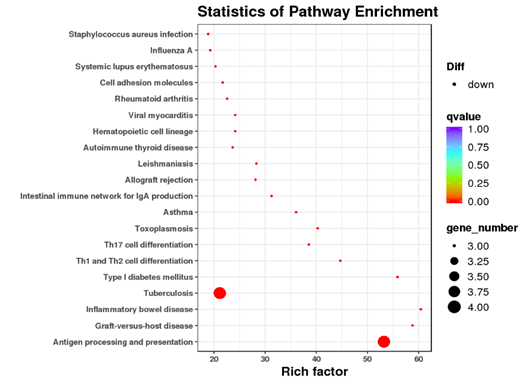
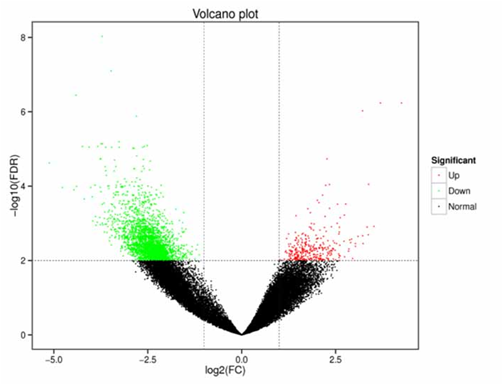
Transcriptomics (RNA-Seq)
De Novo RNA sequencing
De novo RNA sequencing은 Reference Genome이 존재하지 않는 경우 사용하는 RNA-seq 방법으로, 시퀀싱된 Short reads를 이용해 Transcriptome을 처음부터 조립(assembly)하는 기술입니다.
Service Features
| Platform (Library) | Illumina Novaseq (PE150) DNBSEQ-T7 |
|---|---|
| Sample Type | RNA |
| Sequencing Data | 6-10Gb |
| Turnaround Time | ~4 weeks after sample QC |
| Analyzable Species | Prokaryotes Eukaryotes (Poly A enriched, Tissues and Plants, and Insects) |
*RNA prep. 서비스가 필요하신 고객은 상담을 통해 진행 가능합니다.
Service Progress
Data Analysis
유전자의 염기서열, 발현 수준, 대체 스플라이싱(alt. splicing) 등을 파악할 수 있으며, 새로운 유전자나 이소폼을 발견하는 데 유용합니다. 주요 Trinity, SOAPdenovo-Trans, Oases 같은 조립 알고리즘을 사용하며, 비모델 생물(non-model organisms) 연구에 널리 활용됩니다.
Demo Results
| Transcriptome assembly and unigene selection | ||
|---|---|---|
| Length Range | Transcript | Unigene |
| 200-300 | 22 (7.05%) | 20 (14.29%) |
| 300-500 | 46 (14.74%) | 30 (21.43%) |
| 500-1000 | 73 (23.40%) | 27 (19.29%) |
| 1000-2000 | 95 (30.45%) | 31 (22.14%) |
| 2000+ | 76 (24.36%) | 32 (22.86%) |
| Total Number | 312 | 140 |
| Total Length | 462,308 | 185,676 |
| N50 Length | 2,102 | 2,181 |
| Mean Length | 1,481.76 | 1,326.26 |
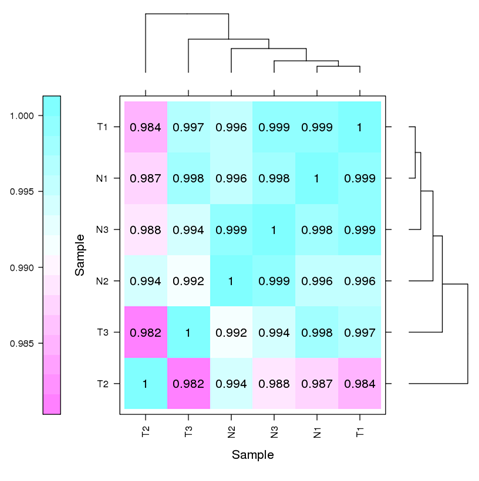
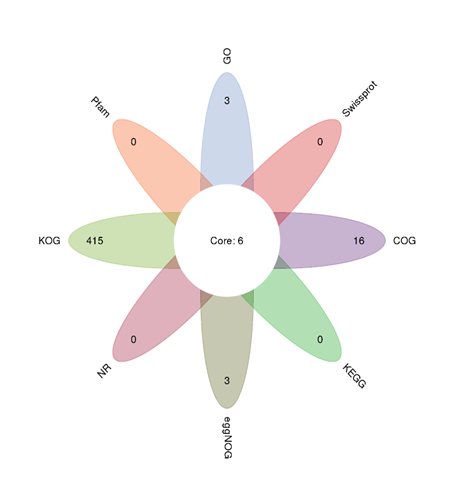
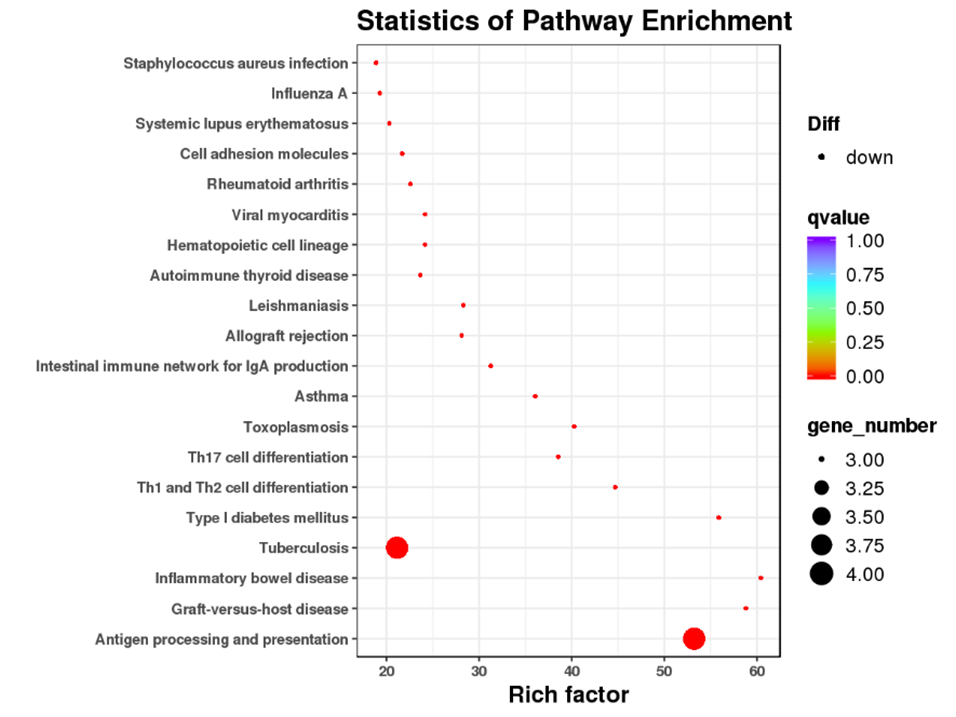
Transcriptomics (RNA-Seq)
Full-Length RNA sequencing
Full-length RNA sequencing은 하나의 전사체(mRNA)의 전체 길이(cDNA 전체)를 읽어내는 시퀀싱 기술로, 유전자 발현뿐 아니라 정확한 전사체 구조(isoform), 스플라이싱 형태, 전사 시작·종결 부위까지 파악할 수 있습니다.
Service Features
| Platform (Library) | Nanopore PromethION 48 PacBio Revio PacBio Sequel II |
|---|---|
| Sample Type | RNA |
| Sequencing Data | 6-40 Gb |
| Turnaround Time | ~6 weeks after sample QC |
| Analyzable Species | Eukaryotes (Poly A enriched, Tissues and Plants) |
*RNA prep. 서비스가 필요하신 고객은 상담을 통해 진행 가능합니다.
Service Progress
Data Analysis
기존의 short-read RNA-seq과 달리 PacBio Iso-Seq이나 Oxford Nanopore와 같은 long-read sequencing 플랫폼을 이용해 조립 없이 완전한 전사체 서열을 직접 얻을 수 있으며, 복잡한 유전자 구조 해석과 새로운 Isoform 탐색에 특히 유용합니다. 2+3 Full Length mRNA Solution을 통해 더욱 완벽한 길이의 RNA Sequencing 수행이 가능합니다.
Demo Results
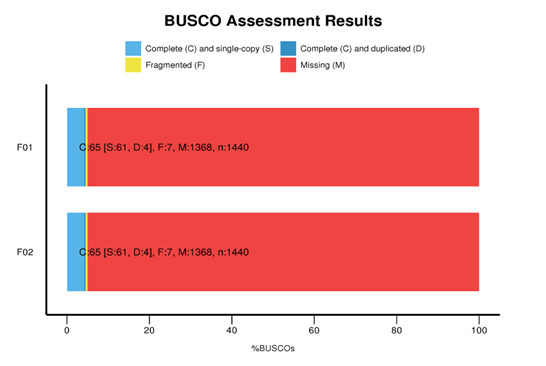
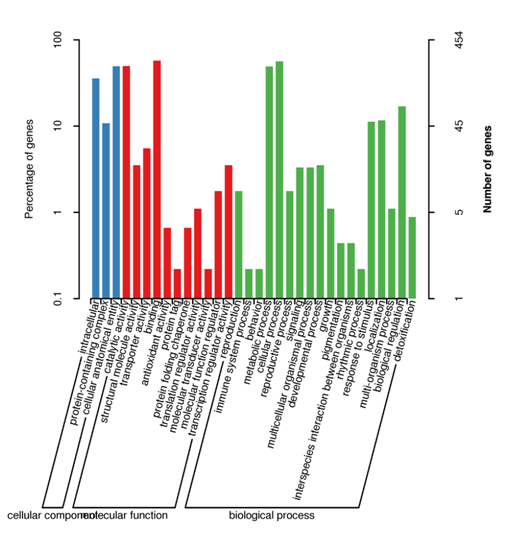
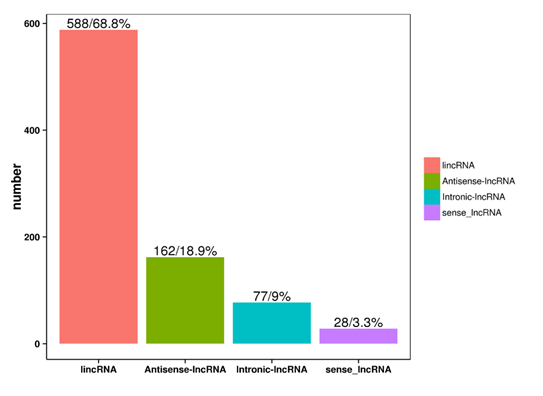
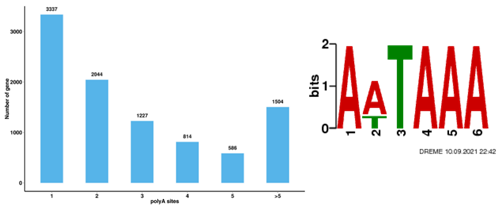
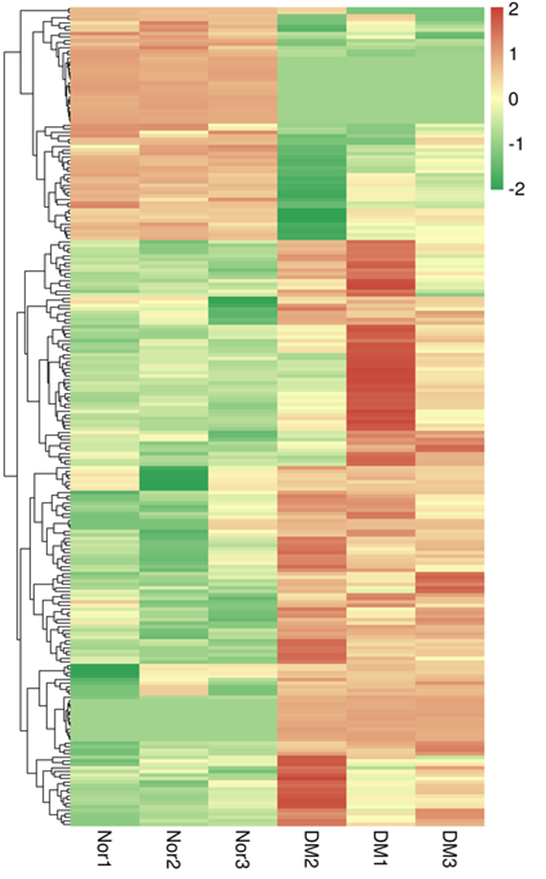
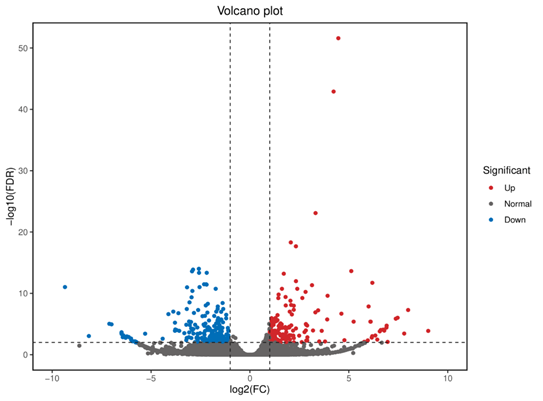
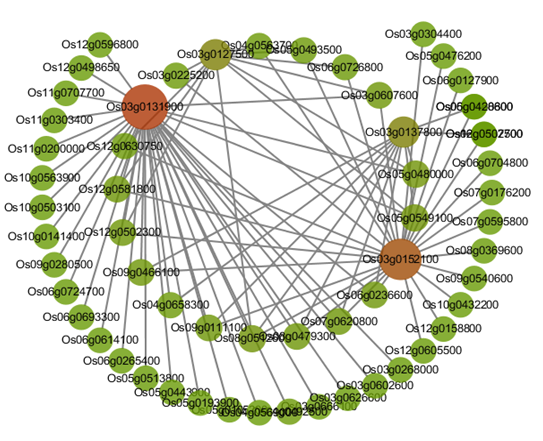
Transcriptomics (RNA-Seq)
LNC/Small/Circular RNA sequencing
Long Non-Coding RNA (lncRNA), Small RNA, 및 Circular RNA (circRNA) 시퀀싱은 단백질을 직접 코딩하지 않지만 유전자 발현에 중요한 역할을 하는 비암호화(non-coding) RNA를 명확히 분석하기 위한 시퀀싱 기법입니다.
Service Features
| Platform (Library) | Illumina Novaseq (PE150, SE150) |
|---|---|
| Sample Type | Non Coding RNA |
| Sequencing Data | 10-20Gb (Size Selected) |
| Turnaround Time | ~4 weeks after sample QC |
| Analyzable Species |
Prokaryotes Eukaryotes (Poly A enriched, Tissues and Plants, and Insects) |
*RNA prep. 서비스가 필요하신 고객은 상담을 통해 진행 가능합니다.
Service Progress
Data Analysis
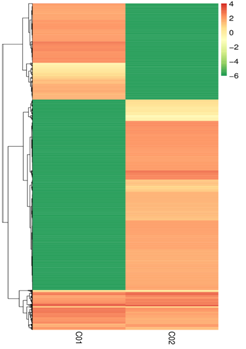
lncRNA-seq은 수천 염기 길이의 긴 비암호화 RNA를 분석해 전사 조절 및 에피제네틱 기능을 연구합니다. Small RNA-seq은 20~30nt 길이의 miRNA, siRNA, piRNA 등을 타깃으로 하며, 유전자 발현 억제나 RNA 간섭 메커니즘을 규명하는 데 활용됩니다. circRNA-seq은 원형 RNA를 탐지하며 스플라이싱 변이 및 유전자 조절 네트워크를 파악합니다. 이 기술은 차세대 시퀀싱(NGS)을 기반으로 하며, 암, 신경질환, 대사질환 등에서 RNA 기반 조절 메커니즘 연구 및 바이오마커 발굴에 활용됩니다.
Demo Results
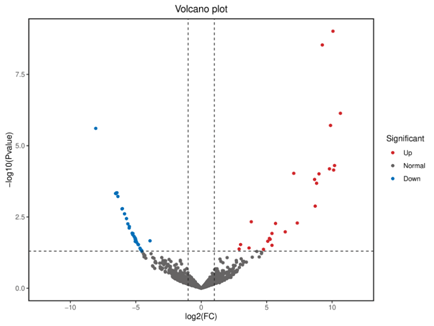
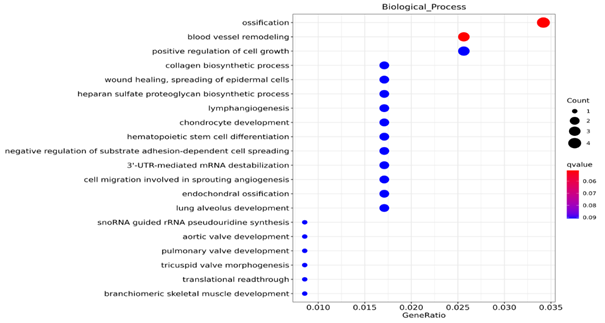
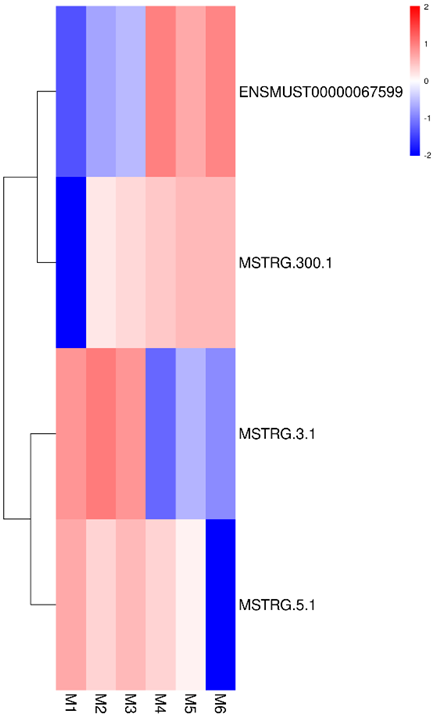
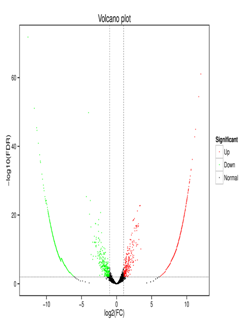
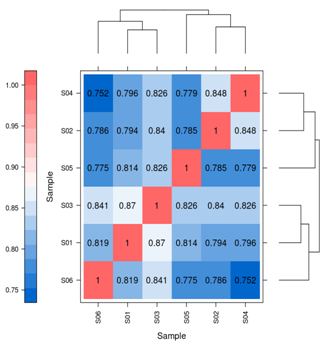
Transcriptomics (RNA-Seq)
Exosomal mRNA/LNC/Circular RNA sequencing
Exosomal lncRNA, mRNA, 및 circular RNA sequencing은 세포가 분비하는 Exosome 내부에 존재하는 다양한 RNA 분자들을 분석하는 시퀀싱 기법입니다.
Service Features
| Platform (Library) | Illumina Novaseq(SE150, PE150) |
|---|---|
| Sample Type | RNA |
| Sequencing Data | 10-20Gb (Size Selected) |
| Turnaround Time | ~4 weeks after sample QC |
| Analyzable Species |
Prokaryotes Eukaryotes (Poly A enriched, Tissues and Plants, and Insects) Liquids (Serum, Plasma, Urine and etc) |
*RNA prep. 서비스가 필요하신 고객은 상담을 통해 진행 가능합니다.
Service Progress
Data Analysis
Exosome은 세포 간 신호 전달을 매개하는 나노 크기의 소포체로, 내부의 lncRNA(긴 비암호화 RNA), mRNA(전령 RNA), circRNA(원형 RNA) 등이 질병 상태나 세포 환경에 따라 변화합니다. 이러한 RNA를 차세대 시퀀싱(NGS)으로 분석하면, 암이나 신경질환 등에서 세포 간 통신, 유전자 조절 메커니즘, 그리고 질병 진단용 바이오마커를 규명할 수 있습니다. 즉, 이 시퀀싱은 비침습적 액체생검(liquid biopsy) 형태로 질병 예측과 정밀의학 연구에 중요한 도구로 활용됩니다.
Demo Results







Transcriptomics (RNA-Seq)
Metatranscriptome Sequencing
Metatranscriptome sequencing은 환경 시료(예: 토양, 해양, 장내 미생물 등)에 존재하는 모든 미생물 군집의 RNA를 추출해 시퀀싱함으로써, Active Gene expression(prokaryotic + eukaryotic)을 총체적으로 분석하는 기법입니다.
Service Features
| Platform (Library) | Illumina Novaseq (PE150) |
|---|---|
| Sample Type | RNA |
| Sequencing Data | 12Gb |
| Turnaround Time | ~4 weeks after sample QC |
| Analyzable Species |
Prokaryotes Eukaryotes (Poly A enriched, Tissues and Plants, and Insects) Liquids (Serum, Plasma, Urine and etc) |
*RNA prep. 서비스가 필요하신 고객은 상담을 통해 진행 가능합니다.
Service Progress
Data Analysis
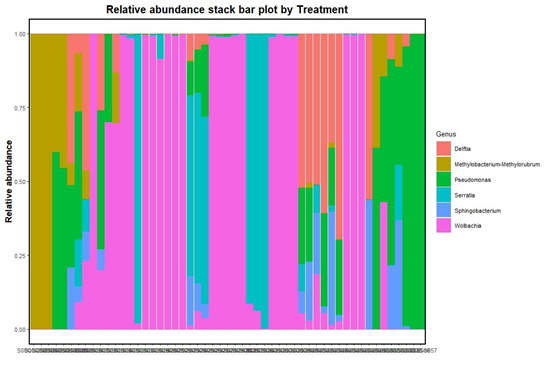
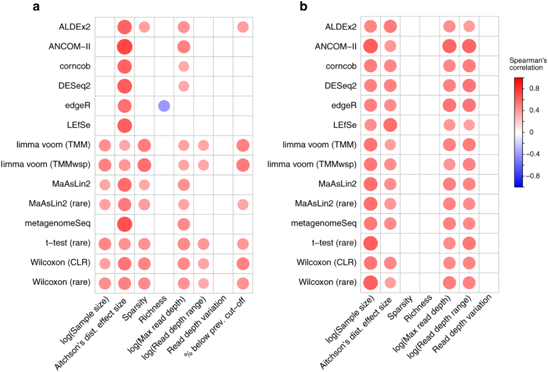
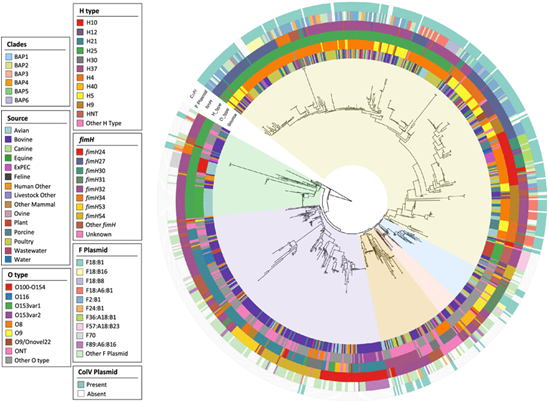
Metagenome sequencing과 달리, 현재 환경 혹은 군집 전체에서 어떤 유전자가 실제로 발현되어 기능하고 있는지를 보여줍니다. 따라서 생태계 내 대사 경로, 상호작용, 환경 반응 메커니즘 등을 규명할 수 있으며, 미생물 군집의 기능적 활성 이해에 핵심적으로 사용됩니다.
Demo Results
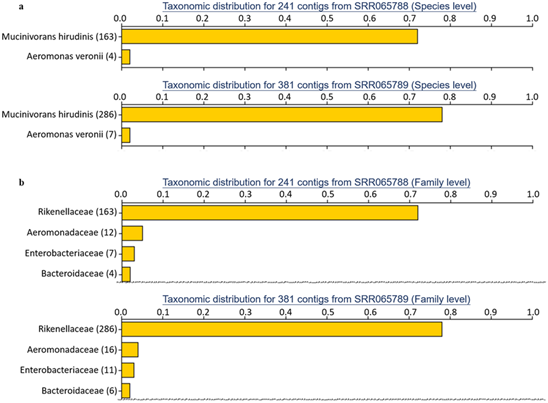
Transcriptomics (RNA-Seq)
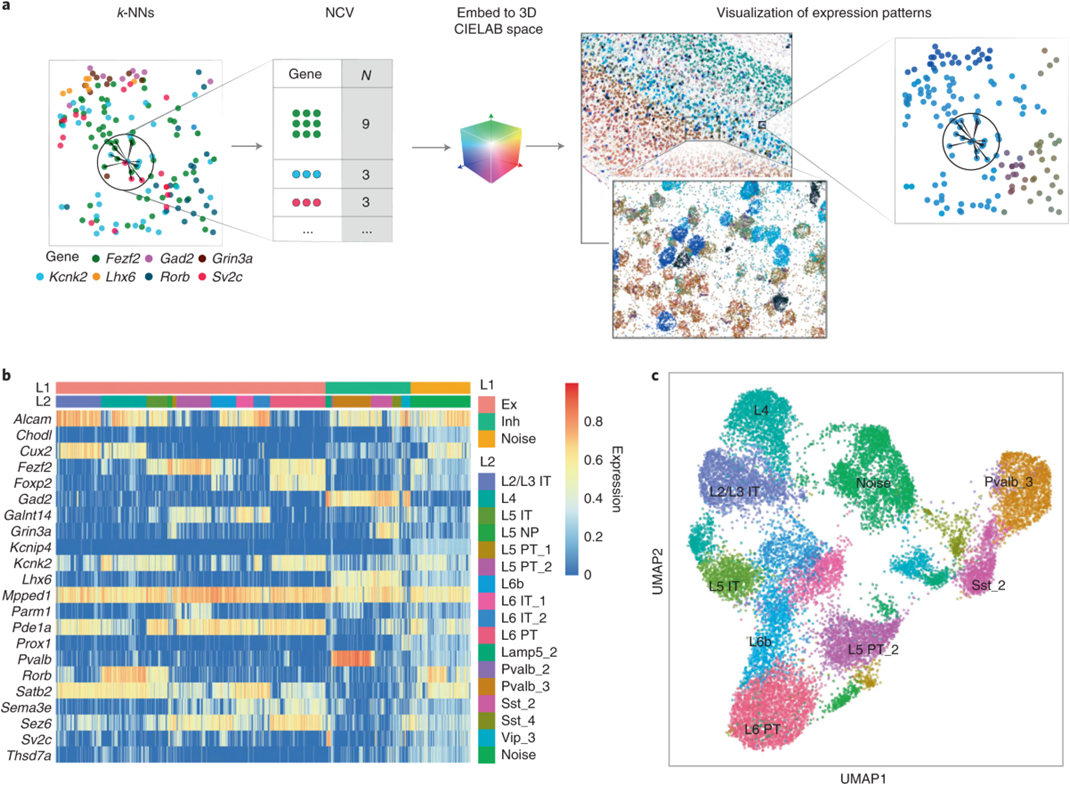
Single-nuclei RNA sequencing
Single-nuclei RNA sequencing (snRNA-seq)은 세포 전체가 아닌 세포핵(nucleus) 단위로 RNA를 추출하여 분석하는 단일세포 전사체 시퀀싱 기법입니다. 세포를 완전히 분리하기 어려운 조직(예: 뇌, 근육, 냉동 시료 등)에서도 핵을 분리해 사용할 수 있어, 손상된 세포나 큰 세포에서도 안정적으로 유전자 발현을 분석할 수 있습니다.
Service Features
| Platform (Library) | Illumina Novaseq (PE150, 10x sn cDNA library) |
|---|---|
| Sample Type | Tissues and cells |
| Sequencing Data | 100K PE per cell (100-200 Gb) |
| Turnaround Time | ~6 weeks after sample QC |
| Analyzable Species |
Neuron cell, Brain, Pancreas, Thyroid, Liver and Muscles and other organs Plants and parts of the plant |
*RNA prep. 서비스가 필요하신 고객은 상담을 통해 진행 가능합니다.
Service Progress
Data Analysis
Single-nuclei RNA sequencing 기법은 세포 유형별 Transcriptome profiling, 희귀 세포군 탐지, 조직 내 세포 다양성 및 발달 경로 분석에 유용하며, 특히 신경과학 및 병리학 연구에서 널리 활용됩니다.
Demo Results
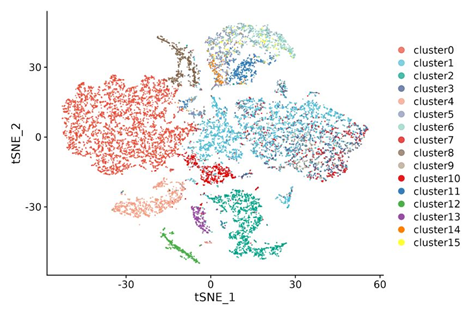
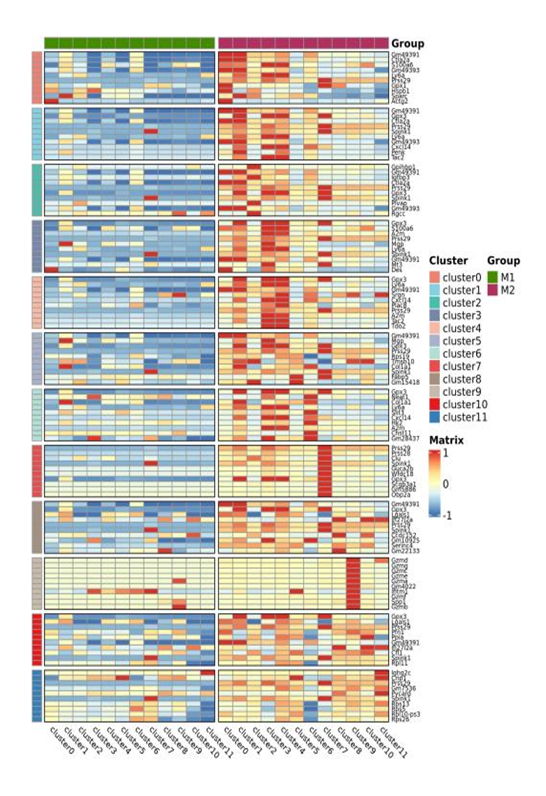
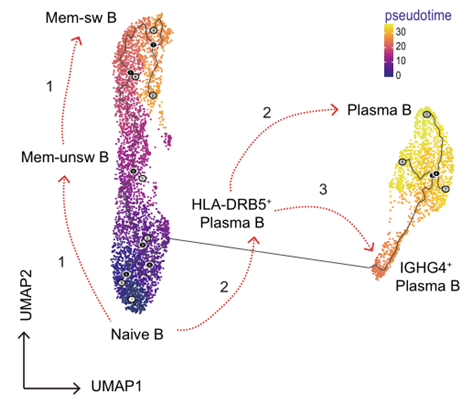
Transcriptomics (RNA-Seq)
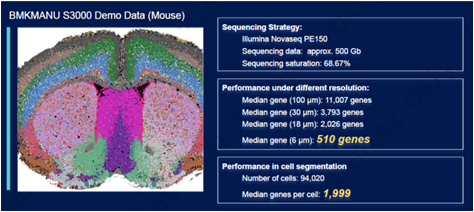
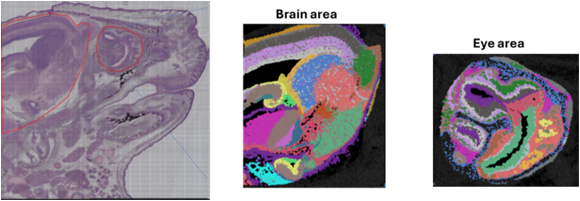
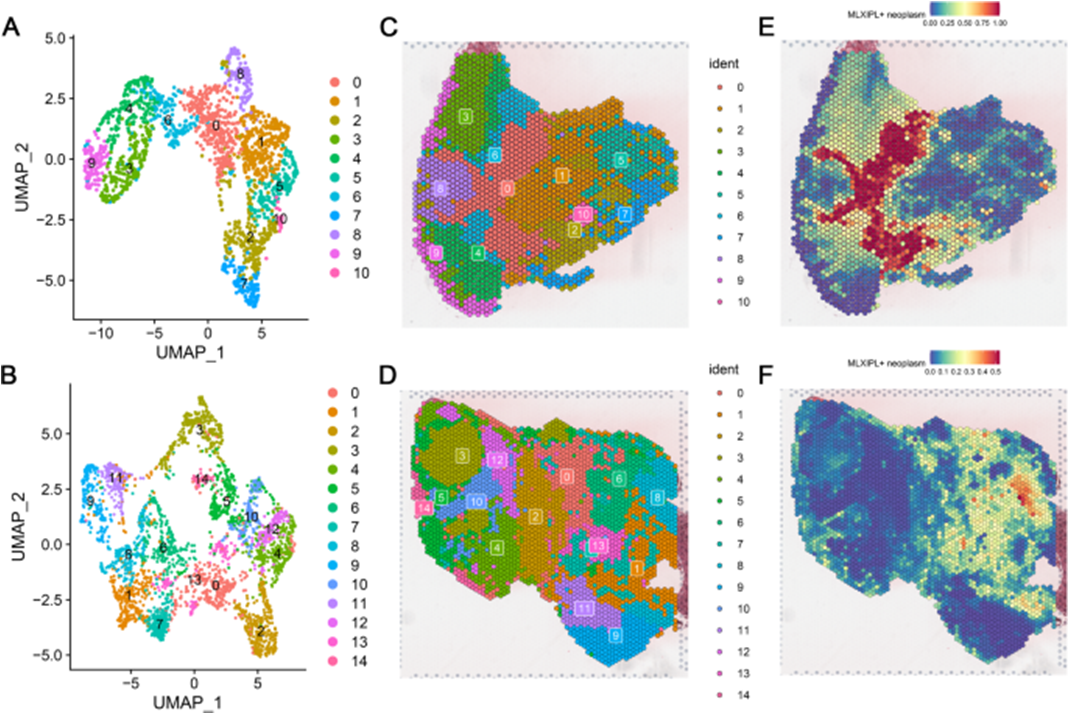
Spatial RNA sequencing
Spatial transcriptomics는 조직 내 공간적 위치 정보(spatial context)를 보존한 상태에서 유전자 발현(RNA expression)을 분석하는 기술입니다. 기존 RNA-seq이 세포의 발현 정보를 평균적으로 제공하는 반면, 이 기법은 어떤 세포가 조직의 어디에서 어떤 유전자를 발현하는지를 시각적으로 보여줍니다.
Service Features
| Platform (Library) |
Illumina Novaseq (PE150, S3000 cDNA library) Illumina Novaseq (PE150, 10X Visium cDNA library) |
|---|---|
| Sample Type | Tissues and cells |
| Sequencing Data |
160K PE per cell (250 Gb) for S3000 library 50K PE reads per spot (60 Gb) for 10X Visium cDNA library |
| Turnaround Time | ~6 weeks after sample QC |
| Analyzable Species |
Neuron cell, Brain, Pancreas, Thyroid, Liver and Muscles and other organs Plants and parts of the plant |
Service Progress
Data Analysis
Spatial transcriptomics 기법을 통해 Tissue structures, Micro environment(예: 중앙 미세환경), 세포 상호작용 등을 정밀하게 규명할 수 있습니다. 대표적인 플랫폼으로는 10x Genomics Visium이 있으며, 병리학·신경과학·암 연구 등에서 활발히 활용됩니다.
Demo Results